本案例为COVID-19疫情的数据科学实践系列的第二篇——Pandas疫情探索性分析。
目录¶
- 数据及Pandas工具介绍
- 实时数据探索性分析
2.1 世界各国实时数据探索性分析
2.2 全国各省实时数据探索性分析 - 历史数据探索性分析
3.1 全国历史数据探索性分析
3.2 世界各国历史数据探索性分析 - 总结
在第一篇案例中我们基于网易实时疫情播报平台,使用Python对疫情数据进行了爬取。本篇案例的主要内容是新冠肺炎疫情数据的探索性分析,包括中国各省和世界各国的实时数据,及中国和世界各国的历史数据。数据集中出现的特征和含义如下表所示:
| 列名 | 含义 |
|---|---|
| date | 日期 |
| name | 名称 |
| id | 编号 |
| lastUpdateTime | 更新时间 |
| today_confirm | 当日新增确诊 |
| today_suspect | 当日新增疑似 |
| today_heal | 当日新增治愈 |
| today_dead | 当日新增死亡 |
| today_severe | 当日新增重症 |
| today_storeConfirm | 当日现存确诊 |
| total_confirm | 累计确诊 |
| total_suspect | 累计疑似 |
| total_heal | 累计治愈 |
| total_dead | 累计死亡 |
| total_severe | 累计重症 |
Pandas是一个开源的、专注于数据分析的Python库。最初由AQR Capital Management于2008年4月开发,并于2009年底开源出来,目前由专注于Python数据包开发的PyData开发小组继续开发和维护,属于PyData项目的一部分。Pandas最初被作为金融数据分析工具而开发出来,因此,Pandas为时间序列分析提供了很好的支持。
Pandas是基于NumPy数组构建的,能够灵活处理关系型数据,可便捷的完成索引、切片、组合以及选取数据子集等操作。接下来就让我们一起使用Pandas对疫情数据进行探索性分析。
我们首先读入数据,将列名英文改为中文。接着,查看数据的基本信息并进行缺失值处理。此外,我们还将新增病死率一列,并将国家设置为索引。数据预处理之后我们将查看世界当前累计确诊人数前十名的国家,并绘制累计确诊、累计死亡和病死率的水平条形图来分析各国疫情状况。
import pandas as pd
# 读取数据
today_world = pd.read_csv("./input/today_world_2020_03_31.csv")
# 查看世界各国实时数据
today_world.head()
数据表各列名称为英文不便于观察,我们将列名修改为中文。首先,创建中英文对照的列名字典,使用rename()函数修改列名:
name_dict = {'date':'日期','name':'名称','id':'编号','lastUpdateTime':'更新时间',
'today_confirm':'当日新增确诊','today_suspect':'当日新增疑似',
'today_heal':'当日新增治愈','today_dead':'当日新增死亡',
'today_severe':'当日新增重症','today_storeConfirm':'当日现存确诊',
'total_confirm':'累计确诊','total_suspect':'累计疑似',
'total_heal':'累计治愈','total_dead':'累计死亡','total_severe':'累计重症'}
# 更改列名
today_world.rename(columns=name_dict,inplace=True) # inplace参数判断是否在原数据上进行修改
today_world.head(3)
当我们拿到一份数据的时候,首先需要观察一下数据的基本信息和特征的统计信息。我们可以使用info()查看数据的基本信息:
# 查看数据基本信息
today_world.info()
查看数据的统计信息可以使用describe()函数:
# 默认只计算数值型特征的统计信息
today_world.describe()
数据中当日新增确诊、疑似、治愈、死亡、重症和当日现存确诊中存在大量缺失值。为了便于观察,我们使用isnull()函数查看缺失值,并结合sum()函数计算缺失值比例。
# 计算缺失值比例
today_world_nan = today_world.isnull().sum()/len(today_world)
# 转变为百分数
today_world_nan.apply(lambda x: format(x, '.1%'))
我们发现当日新增相关数据缺失值较多,这主要由于采集数据的当天一些国家没有更新数据,因此我们将不再对其进行分析。当日现存确诊一列虽然全部为空,但该缺失值可以通过已有数据直接计算,公式为:
$$当日现存确诊=累计确诊-累计治愈-累计死亡$$# 缺失值处理
today_world['当日现存确诊'] = today_world['累计确诊']-today_world['累计治愈']-today_world['累计死亡']
除了我们数据中提供的这些特征,病死率也是一个非常重要的特征,能够反映疾病的严重程度以及一个地区的医疗水平。接下来,我们来看看截止数据采集当天,各国的病死率情况。病死率的计算公式为:
$$病死率=累计死亡\div累计确诊$$# 计算病死率,且保留两位小数
today_world['病死率'] = (today_world['累计死亡']/today_world['累计确诊']).apply(lambda x: format(x, '.2f'))
# 将病死率数据类型转换为float
today_world['病死率'] = today_world['病死率'].astype('float')
# 根据病死率降序排序
today_world.sort_values('病死率',ascending=False,inplace=True)
# 显示病死率前十国家
today_world.head(10)
排名第一的国家苏丹病死率高达0.29,但从表中可知,该国累计确诊人数只有7例。可见,病死率应结合累计确诊人数一起查看。
为了方便查询特定国家的数据,我们使用set_index()函数将国家设置为索引:
# 将国家名称设为索引
today_world.set_index('名称',inplace=True)
today_world.head(3)
today_world.loc['中国'] #可以通过传入列表获取多个国家的数据
当前累计确诊人数top10国家
接下来,让我们使用sort_values()函数根据累计确诊人数进行排序,找到确诊人数前十国家的累计确诊、累计死亡、病死率三列数据:
# 查看当前累计确诊人数前十国家
world_top10 = today_world.sort_values(['累计确诊'],ascending=False)[:10]
world_top10 = world_top10[['累计确诊','累计死亡','病死率']]
world_top10
直接观察数据并不直观,我们需要借助可视化方法来进行分析。
# 导入matplotlib
import matplotlib.pyplot as plt
plt.rcParams['font.sans-serif']=['SimHei'] #正常显示中文
plt.rcParams['figure.dpi'] = 120 #设置所有图片的清晰度
绘制当前累计确诊人数top10国家的累计确诊、累计死亡和病死率的水平条形图。
# 绘制条形图
world_top10.sort_values('累计确诊').plot.barh(subplots=True,layout=(1,3),sharex=False,
figsize=(7,4),legend=False,sharey=True)
plt.tight_layout() #调整子图间距
plt.show()
由图可知,目前美国累计确诊病例已远远高于其他国家,意大利、西班牙人数也相对较高。而在病死率的图表上意大利则位列第一,可见疫情的严重性,西班牙、法国、伊朗、英国、比利时病死率紧跟其后。然而,累计确诊人数最高的美国病死率却相对较低,其中一个原因就是美国的医疗资源丰富,下图是一张每十万人ICU病床数量的国家排名条形图:
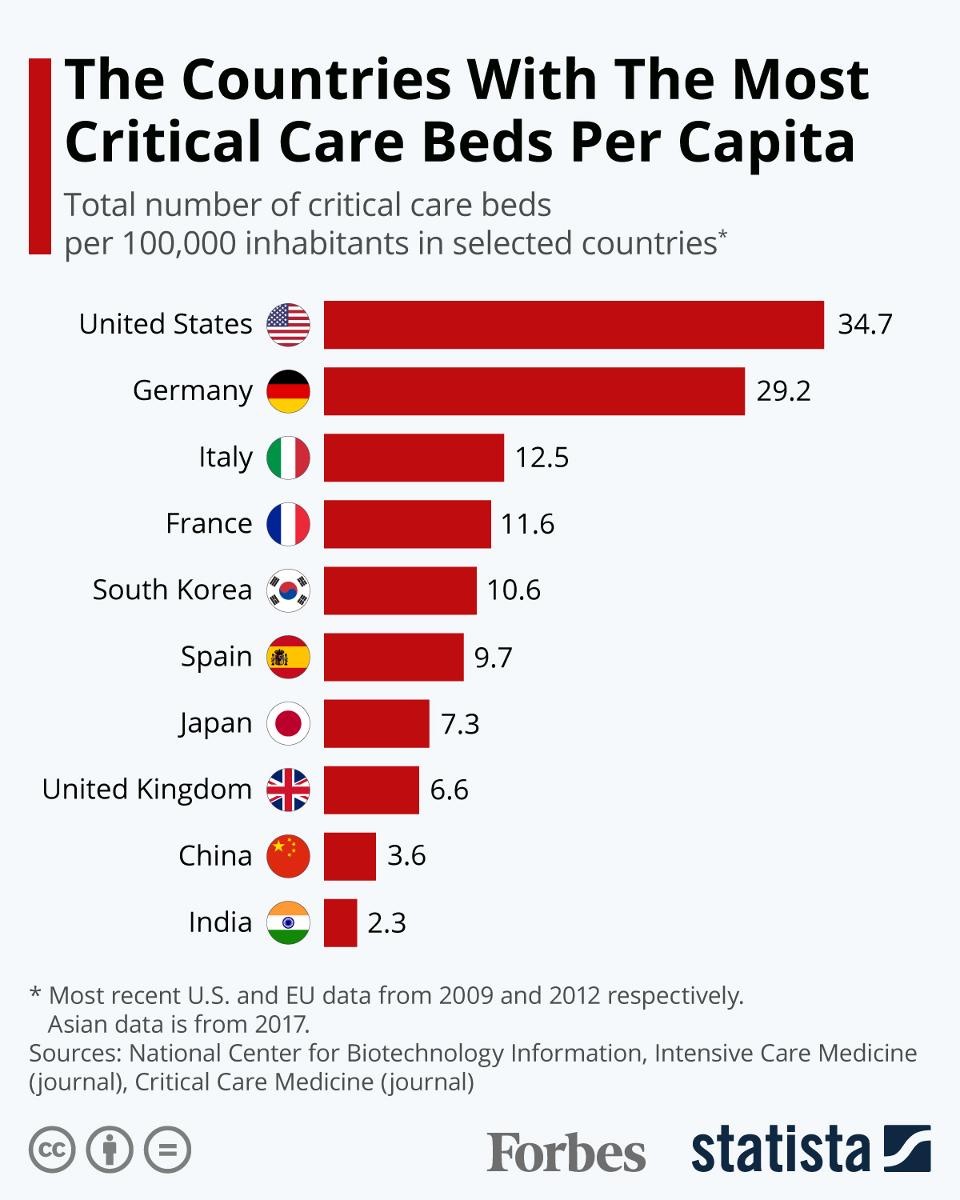
我们发现,美国每十万居民拥有将近35个ICU床位,排在世界第一,德国紧跟其后有近30个ICU床位,两国的医疗资源远远高于其他国家,这也是新冠肺炎疫病死率相对较低的原因之一。
下面让我们一起来分析国内的新冠肺炎疫情情况。全国各省实时数据的预处理工作与前面的流程基本一致,数据清洗之后我们将分别探索全国新增确诊top10地区,及全国现存确诊人数top10的地区。
# 读取数据
today_province = pd.read_csv("./input/today_province_2020_03_31.csv")
# 创建中文列名字典
name_dict = {'date':'日期','name':'名称','id':'编号','lastUpdateTime':'更新时间',
'today_confirm':'当日新增确诊','today_suspect':'当日新增疑似',
'today_heal':'当日新增治愈','today_dead':'当日新增死亡',
'today_severe':'当日新增重症','today_storeConfirm':'当日现存确诊',
'total_confirm':'累计确诊','total_suspect':'累计疑似',
'total_heal':'累计治愈','total_dead':'累计死亡','total_severe':'累计重症'}
# 更改列名
today_province.rename(columns=name_dict,inplace=True) # inplace参数是否在原对象基础上进行修改
today_province.head()
# 查看数据基本信息
today_province.info()
使用describe()查看数据的统计信息:
# 查看数值型特征的统计量
today_province.describe()
我们发现,累计疑似、累计重症、当日新增疑似和当日新增重症的数据全部为0,因此我们便不予考虑。
# 计算各省当日现存确诊人数
today_province['当日现存确诊'] = today_province['累计确诊']-today_province['累计治愈']-today_province['累计死亡']
# 将各省名称设置为索引
today_province.set_index('名称',inplace=True)
today_province.info()
全国新增确诊top10地区
目前,我国疫情已得到很好的控制,我们现在更为关注的是新增确诊病例的地区。
# 查看全国新增确诊top10的地区
new_top6 = today_province['当日新增确诊'].sort_values(ascending=False)[:10]
new_top6
# 绘制条形图和饼图
fig,ax = plt.subplots(1,2,figsize=(10,5))
new_top6.sort_values(ascending=True).plot.barh(fontsize=10,ax=ax[0])
new_top6.plot.pie(autopct='%.1f%%',fontsize=10,ax=ax[1])
plt.ylabel('')
plt.title('全国新增确诊top10地区',size=15)
plt.show()
从图中可知,香港、台湾新增确诊人数最多,且在新增确诊前十名的地区,香港占比将近一半。
全国现存确诊人数top10的地区
接下来我们查看一下全国现存确诊病例前十名的地区有哪些。
# 查看全国现存确诊人数top10的省市
store_top10 = today_province['当日现存确诊'].sort_values(ascending=False)[:10]
store_top10
# 绘制条形图
store_top10.sort_values(ascending=True).plot.barh(fontsize=10)
plt.title('全国现存确诊top10地区',size=15)
plt.show()
虽然湖北现存确诊人数仍位居第一,但已鲜有新增确诊病例。
全国历史数据是时间序列的数据类型,在数据清洗的时候需要对时间进行处理。本部分的最后我们将绘制全国历史数据的折线图,并着重分析全国新增确诊人数的变化趋势。
# 读取数据
alltime_china = pd.read_csv("./input/alltime_China_2020_03_31.csv")
# 创建中文列名字典
name_dict = {'date':'日期','name':'名称','id':'编号','lastUpdateTime':'更新时间',
'today_confirm':'当日新增确诊','today_suspect':'当日新增疑似',
'today_heal':'当日新增治愈','today_dead':'当日新增死亡',
'today_severe':'当日新增重症','today_storeConfirm':'当日现存确诊',
'total_confirm':'累计确诊','total_suspect':'累计疑似',
'total_heal':'累计治愈','total_dead':'累计死亡','total_severe':'累计重症'}
# 更改列名
alltime_china.rename(columns=name_dict,inplace=True)
alltime_china.head()
alltime_china.info()
# 查看数据的统计信息
alltime_china.describe()
# 缺失值处理
# 计算当日现存确诊人数
alltime_china['当日现存确诊'] = alltime_china['累计确诊']-alltime_china['累计治愈']-alltime_china['累计死亡']
# 删除更新时间一列
alltime_china.drop(['更新时间','当日新增重症'],axis=1,inplace=True)
alltime_china.info()
与实时数据相比,历史数据的日期一列是非常重要的。我们使用pd.to_datetime()将日期的数据类型设为datetime,并将其设置为行索引。
# 将日期改成datetime格式
alltime_china['日期'] = pd.to_datetime(alltime_china['日期'])
# 设置日期为索引
alltime_china.set_index('日期',inplace=True) # 也可使用pd.read_csv("./input/alltime_China_2020_03_27.csv",parse_dates=['date'],index_col='date')
alltime_china.index
设置为时期索引后,数据的选取将非常便利。
# 举例
alltime_china.loc['2020-01']
数据清洗之后,我们将绘制折线图查看新冠肺炎数据的变化趋势:
# 时间序列数据绘制折线图
import matplotlib.pyplot as plt
import matplotlib.dates as dates
import matplotlib.ticker as ticker
import datetime
fig, ax = plt.subplots(figsize=(8,4))
alltime_china.plot(marker='o',ms=2,lw=1,ax=ax)
ax.xaxis.set_major_locator(dates.MonthLocator()) #设置间距
ax.xaxis.set_major_formatter(dates.DateFormatter('%b')) #设置日期格式
fig.autofmt_xdate() #自动调整日期倾斜
# 图例位置调整
plt.legend(bbox_to_anchor = [1,1])
plt.title('全国新冠肺炎数据折线图',size=15)
plt.ylabel('人数')
plt.grid(axis='y')
plt.box(False)
plt.show()
由图可知,我国累计确诊人数在2月中旬已达到拐点,现存确诊人数也已从2月15日起逐步减少。同时,累计治愈人数稳步上升,且随现存确诊人数的下降而逐渐趋于平缓状态。由于新增确诊等字段数值相对较小,我们单独进行分析。
# 时间序列数据绘制折线图
fig, ax = plt.subplots(figsize=(8,4))
alltime_china['当日新增确诊'].plot(ax=ax, style='-',lw=1,color='c',marker='o',ms=3)
ax.xaxis.set_major_locator(dates.MonthLocator()) #设置间距
ax.xaxis.set_major_formatter(dates.DateFormatter('%b')) #设置日期格式
fig.autofmt_xdate() #自动调整日期倾斜
plt.title('全国新冠肺炎新增确诊病例折线图',size=15)
plt.ylabel('人数')
plt.grid(axis='y')
plt.box(False)
plt.show()
可以看到2月12日新增病例大幅上升,这是什么原因呢?如果你非常关注疫情,应该知道这是因为在当天国家卫健委调整了确诊的标准。此前,患者是否确诊,主要参考指标是核酸检测结果,还需要结合CT影像,咳嗽等症状来综合判断。随着疫情防控深入,临床数据的不断累积,有个新情况逐渐凸显出来——由于核酸检测的时间较慢,一些患者无法确诊收治,但是病症的临床表现又高度疑似新冠肺炎。如果不改变认定标准,这部分患者就难以得到有效救助,对整个疫情防控也造成负面影响。这次主要变化,就是将临床诊断纳入确诊范围。
最后让我们一起来分析一下世界各国历史数据,由于数据表中每个国家含有多条数据,我们需要借助GroupBy技术对数据进行分组,并通过层次化索引操作选取多个国家的累计确诊和新增确诊数据,来查看各国疫情变化趋势。
# 读取数据
alltime_world = pd.read_csv("./input/alltime_world_2020_03_31.csv")
# 创建中文列名字典
name_dict = {'date':'日期','name':'名称','id':'编号','lastUpdateTime':'更新时间',
'today_confirm':'当日新增确诊','today_suspect':'当日新增疑似',
'today_heal':'当日新增治愈','today_dead':'当日新增死亡',
'today_severe':'当日新增重症','today_storeConfirm':'当日现存确诊',
'total_confirm':'累计确诊','total_suspect':'累计疑似',
'total_heal':'累计治愈','total_dead':'累计死亡','total_severe':'累计重症'}
# 更改列名
alltime_world.rename(columns=name_dict,inplace=True)
alltime_world.head()
# 查看数据基本信息
alltime_world.info()
alltime_world.describe()
数据预处理操作与前面的方法基本一致:
# 将日期一列数据类型变为datetime
alltime_world['日期'] = pd.to_datetime(alltime_world['日期'])
# 计算当日现存确诊
alltime_world['当日现存确诊'] = alltime_world['累计确诊']-alltime_world['累计治愈']-alltime_world['累计死亡']
alltime_world.info()
数据表中总共有哪些国家呢?我们可以使用unique()查看数据中的唯一值:
# 查看唯一值,可使用len()查看个数
alltime_world['名称'].unique()
同时我们还想了解随着时间的变化,每天有多少国家出现新冠肺炎疫情,value_counts()函数可帮助我们查看每一天记录了多少数据。
# 统计每天有多少国家出现疫情
alltime_world['日期'].value_counts().head(20)
数据中显示,3月26日这天出现疫情的国家数量已多达157个国家。
# 设置日期索引
alltime_world.set_index('日期',inplace=True)
# 3月31日数据统计不完全,我们将其删除
alltime_world = alltime_world.loc[:'2020-03-31']
选取多国数据
接下来,我们想提取中国、日本、韩国、美国、意大利、英国、西班牙和德国的数据,探索这八个国家的累计确诊和新增确诊病例的变化趋势。我们将使用GroupBy技术和层次化索引操作。GroupBy技术是对数据进行分组计算并将各组计算结果合并的一项技术,包括如下三个过程:
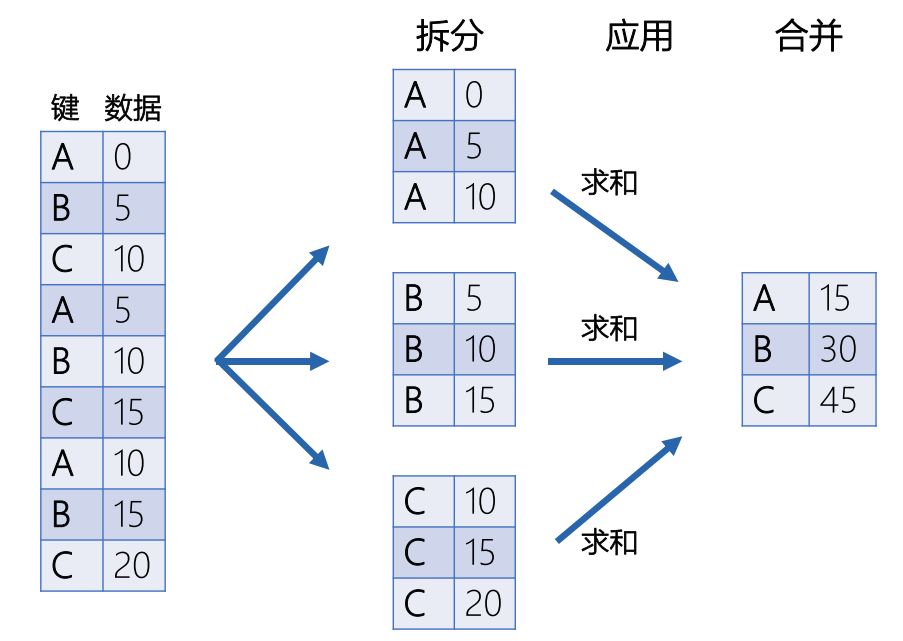
想要提取多个国家的数据,就需要把国家一列也设置为索引,我们可以使用groupby()函数根据日期和名称两列进行分组,将数据转为层次化索引。
# groupby创建层次化索引
data = alltime_world.groupby(['日期','名称']).mean()
data.head()
想要提取部分数据,同样可以使用.loc方法,需先通过.loc(axis= )指定对行索引还是对列索引进行操作。比如,我们想提取中国、韩国、美国、意大利、英国、西班牙、德国的数据:
# 提取部分数据
data_part = data.loc(axis=0)[:,['中国','日本','韩国','美国','意大利','英国','西班牙','德国']]
data_part.head()
此时,多级索引已设置成功。如果我们想将其还原则可使用reset_index()函数。
# 将层级索引还原
data_part.reset_index('名称',inplace=True)
data_part.head()
绘制多个国家的累计确诊人数折线图
# 绘制多个国家的累计确诊人数折线图
fig, ax = plt.subplots(figsize=(8,4))
data_part['2020-02':].groupby('名称')['累计确诊'].plot(legend=True,marker='o',ms=3,lw=1)
ax.xaxis.set_major_locator(dates.MonthLocator()) #设置间距
ax.xaxis.set_major_formatter(dates.DateFormatter('%b')) #设置日期格式
fig.autofmt_xdate() #自动调整日期倾斜
plt.title('各国新冠肺炎累计确诊病例折线图',size=15)
plt.ylabel('人数')
plt.grid(axis='y')
plt.box(False)
plt.legend(bbox_to_anchor = [1,1])
plt.show()
由图可知,中韩两国目前累计确诊人数已出现拐点,疫情趋于平稳,而美国在最近几天累计确诊人数呈现爆发性的增长,已位居世界第一。此外,意大利、西班牙两国增长速度也紧跟其后。
最后,让我们一起来观察一下各国新增确诊人数的变化情况,这里我们只查看3月份的最新数据。
# 绘制各国新增确诊人数折线图
fig, ax = plt.subplots(figsize=(8,4))
data_part['2020-03':'2020-03-29'].groupby('名称')['当日新增确诊'].plot(legend=True,marker='o',ms=3,lw=1)
ax.xaxis.set_major_locator(dates.MonthLocator()) #设置间距
ax.xaxis.set_major_formatter(dates.DateFormatter('%b')) #设置日期格式
fig.autofmt_xdate() #自动调整日期倾斜
plt.title('各国新冠肺炎新增确诊病例折线图',size=15)
plt.ylabel('人数')
plt.grid(axis='y')
plt.box(False)
plt.legend(bbox_to_anchor = [1,1])
plt.show()
各国新增确诊人数波动较大,但总体趋势呈上升状态。在3月下旬,美国和西班牙首次单日新增确诊人数破万,而最新数据显示美国单日新增已突破25000例。
在前面两张图里,我们发现日本由于数据较小,很难观察疫情的变化趋势。这里我们单独选取了日本新冠肺炎的累计确诊和当日新增确诊两列数据进行绘制:
japan = alltime_world[alltime_world['名称']=='日本']
fig, ax = plt.subplots(figsize=(8,4))
japan['累计确诊'].plot(ax=ax, fontsize=10, style='-',lw=1,color='c',marker='o',ms=3,legend=True)
ax.set_ylabel('人数', fontsize=10)
ax1 = ax.twinx()
ax1.bar(japan.index, japan['当日新增确诊'])
ax1.xaxis.set_major_locator(dates.DayLocator(interval = 5))
ax1.xaxis.set_major_formatter(dates.DateFormatter('%b %d'))
ax1.legend(['当日新增确诊'],loc='upper left',bbox_to_anchor=(0.001, 0.9))
plt.grid(axis='y')
plt.box(False)
plt.title('日本新冠肺炎疫情折线图',size=15)
plt.show()
我们发现日本前期新增确诊人数的变化几乎没有太大增幅,但在3月25日起,日本的新增确诊人数明显增大,同时累计确诊折线斜率也随之增加。
本案例使用基于网易实时疫情播报平台爬取的数据,进行新冠肺炎疫情数据的探索性分析。其中数据预处理主要包括特征列重命名、缺失值处理、查看重复值、数据类型转换等操作。此外,我们还使用了Pandas进行数据可视化,通过图表的绘制探索数据的内涵。同时,我们介绍了时间序列数据的处理方法、如何使用Groupby技术进行数据分组,以及层次化索引的操作方法。